

Ako sa hľadajú a vyvíjajú dátové DNA úložiská
Kam a ako budeme dáta ukladať za desať či päťdesiat rokov? Aké nové metódy ukladania v súčasnosti preskúmavame a aké pokroky sme v posledných rokoch dosiahli? A hlavne, aké prekážky musíme ešte prekonať, než sa tieto nové technológie zhmotnia a preberú dátové opraty?
V súčasnosti digitálne dáta ukladáme na úložiská tromi základnými spôsobmi. Nabíjaním tranzistorov a čítaním ich elektrickej hodnoty (čo je prípad NAND flash a RAM), zmenou orientácie magnetických regiónov (HDD, dátové pásky) a fyzických dierkami, ktoré strojovo lisujeme alebo vypaľujeme laserom (Blu-ray, DVD, CD). Spôsoby, akými tieto veci robíme, sa pravdaže neustále vyvíjajú a pokrok v technológiách nám umožňuje vyrábať z hľadiska kapacity stále väčšie a dátovo hustejšie úložiská. Z pár kilobajtových NAND flash čipov (v SSD diskoch či smartfónoch) sa tak behom dekád vyvinuli niekoľko gigabajtové a z pár megabajtových pevných diskov zas niekoľko terabajtové. Všetko hlavne zmenšovaním ich hlavných častí v podobe tranzistorov a magnetických zŕn. To platí aj pre optické médiá v podobe DVD a Blu-ray a zmenšovanie ich dierok, čo je prakticky len pokračovaním princípu štítkov a pások z ranných dôb počítačov, keď sa dierky razili do papiera. Či už ide o elektrické nabitie hradla tranzistora, alebo orientáciu magnetických zŕn, na ukladanie dát obvykle používame zmenu vlastností nejakého materiálu. V základe je však hon na hustotu zápisu honom na to, aby jednotku informácie (bit) niesol čo najmenší prvok. Ideálnym stavom by bolo, ak by kompletnú dátovú hodnotu niesla len jediná molekula, teda najmenšia častica konkrétnej látky (podľa typu zložená z dvoch alebo viacerých atómov), ktorá má ešte svoje kompletné chemické vlastnosti. Molekulárne úložiská sú z tohto hľadiska atraktívne a aj keď existuje mnoho rôznych konfigurácií ako ich dosiahnuť, tou suverénne najlákavejšou je DNA.
Prečo nás DNA úložiská lákajú
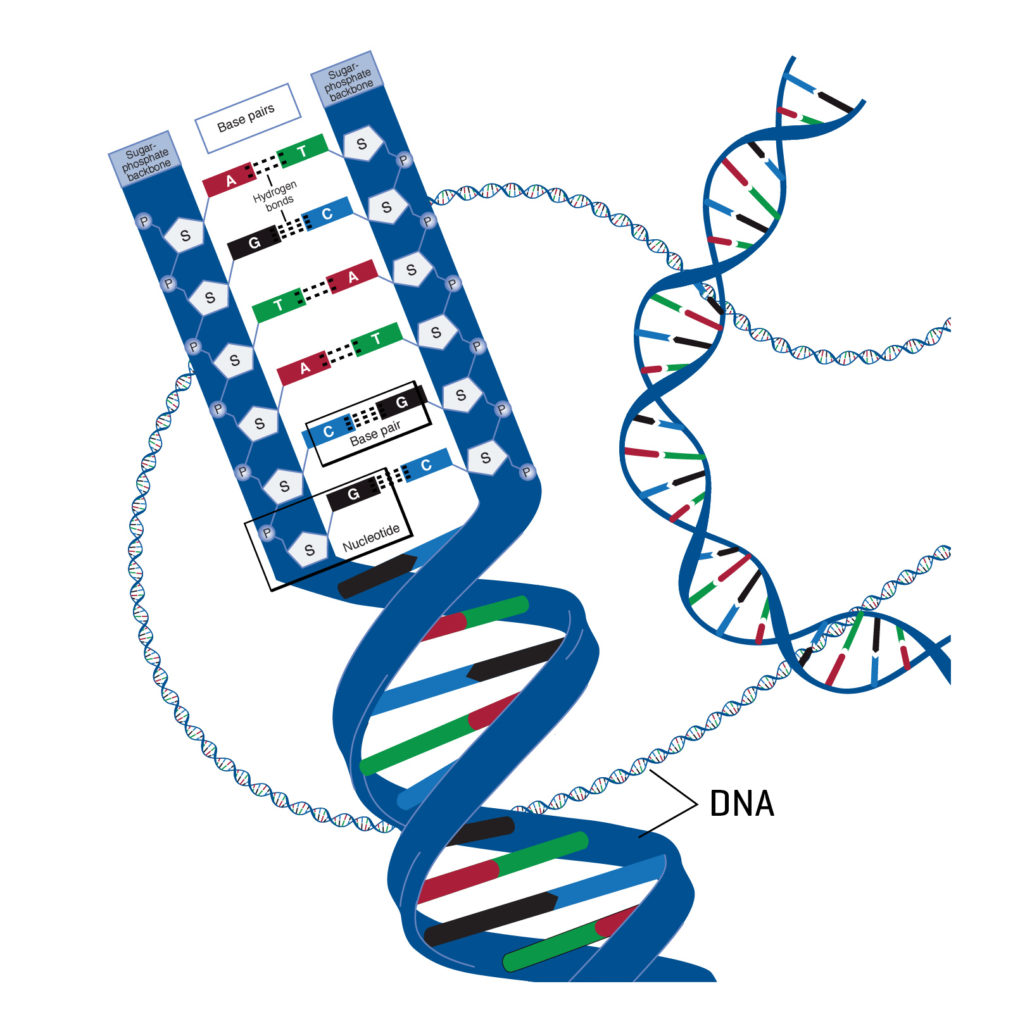
DNA je najhustejšie a najpreverenejšie dátového úložisko, aké poznáme. Príroda ho vyvinula evolučnou úpravou RNA a iných chemických mechanizmov už pred niekoľkými miliardami rokov, pričom je základným stavebným kameňom všetkého súčasného života na zemi. Nemusíme premýšľať nad tým, či tento typ molekulárneho úložiska teoreticky zvládne uchovávať veľké objemy dát a či ich udrží aj na dlhú dobu, pretože to vidíme v praxi potvrdené v každom živom organizme, vrátane nás samých. Táto preverená funkčnosť je teda z pohľadu vývoja digitálnych úložísk veľmi lákavá. DNA sa v základe skladá zo štyroch rôznych látok, tzv. nukleotidov, ktoré pre zjednodušenie nazývame písmená A, C, G a T. V skutočnosti pravdaže o žiadne písmená nejde a za týmto označením sa skrývajú organické chemikálie. Ide o dusíkaté bázy v podobe adenínu (A), guanínu (G), cytozínu (C) a tymínu (T). Striedaním týchto nukleotidov je možné uchovávať informáciu, podobne ako to robíme striedaním rôzne nabitých tranzistorov, alebo rôzne zmagnetizovaných skupín zŕn (juh/sever), čo reprezentuje jednotky a nuly na súčasných úložiskách.
DNA je najhustejšie a najpreverenejšie dátového úložisko, aké poznáme.
Do očí udierajúcim faktom je, že jeden nukleotid má rozmer len 0,33 nanometra, pričom sa v rámci DNA sám páruje s inými nukleotidmi a zamotáva do známej dvojitej špirály. Ľudská DNA sa skladá zo zhruba 3 miliárd takýchto párov, pričom pri rozmere 0,6 nm na každý pár sa dostávame na dĺžku špirály zhruba 1,8 metra. To na prvý pohľad môže pôsobiť veľa, obzvlášť ak to v rečiach digitálnych dát znamená len zhruba 3 GB. Avšak, keďže priemer špirály je len 2,2 až 2,6 nanometra, tak vďaka svojej tenkosti, flexibilite a zamotaniu je bez problémov „zaklbkovaná“ prakticky v každej našej bunke (0,001 mm). To je skoro až neuveriteľná dátová hustota na úrovni 1 exabajtu (miliónu terabajtov) na jeden milimeter kubického objemu. Od takýchto čísiel sú súčasné najpokročilejšie úložiská v podobe HDD a SSD vzdialené o šesť rádov, čo zodpovedá rozdielu veľkosti zrnka piesku od planéty Zem.
Inak povedané, ak by sme vedeli dáta do DNA zapísať a uchovať najúčinnejším možným spôsobom, všetky súčasné digitálne dáta ľudstva (33 zettabajtov) by sa zmestili do hrnčeka na rannú kávu. Tu je vhodné ale upozorniť, že ide o potenciál. Neznamená to, že prevádzka takéhoto dátového úložiska bude vôbec niekedy prakticky možná, a že sa k takémuto úplnému využitiu niekedy vôbec priblížime. Ak by sa však podarilo využiť hoc aj 1 % z tohto potenciálu, stále by šlo o enormne účinné úložisko. Koniec koncov, 33 zettabajtov je náhodou aj približný objem duplicitných DNA dát, ktoré sa nachádzajú v ľudskom tele (3 GB vo všetkých 30 biliardách, teda tridsiatich miliónoch miliónov buniek).
Všetky súčasné digitálne dáta ľudstva (33 zettabajtov) by sa zmestili do hrnčeka na rannú kávu
Reálnym problémom akýchkoľvek úložísk je, že aj keď majú dlhú životnosť dát, technologická životnosť môže spôsobiť, že jedného dňa ich už nebudeme mať na čom prečítať. Typicky si spomeňte na dierne štítky či diskety. V priebehu času tak musíme dáta zo starých nosičov presúvať na nové, aj keď nehrozí ich strata z dôvodu fyzickej životnosti, pretože tá technologická nás o možnosť ich čítania pripraví ako prvá. To je potenciálne možné v rámci DNA úložísk prekonať, pretože aj keď sa naše DNA úložiská budú nepochybne meniť a vyvíjať, ich výsledný produkt je stále len stará známa DNA špirála. A recept na jej čítanie bude stále rovnaký a bude na našej planéte dostupný tak dlho, ako na našej planéte bude existovať život.
Na akom princípe DNA úložiská fungujú a ako sme ich začali vyrábať
V DNA môžu byť uchovávané akékoľvek digitálne dáta. Vyžadujú sa na to tie isté kroky, ako pri iných úložiskách. Dáta v binárnej podobe sa prekonvertujú elektronicky najprv na ich fyzické reprezentácie, teda v tomto prípade písmená A, C, G, T. Podľa nich sa následne vytvoria príslušné chemikálie/nukleotidy a zamotajú sa v správnom poradí do DNA špirály. Pri procese čítania sa z nej zas rozmotajú, zistené chemikálie odhalia aké písmená reprezentujú, pričom daný „text“ zas prevedieme na binárne dáta. V princípe teda ide o podobný proces, ako keď sa pri NAND flash binárne dáta prevádzajú na hodnoty elektrického napätia tranzistorov, alebo pri HDD zas na orientáciu magnetických zŕn na sever alebo juh. To, čo je pravdaže komplikované, je samotné fyzické narábanie s nukleotidmi, teda ich správne spájanie a rozkladanie.
Pri týchto postupoch sa používa tzv. sekvencovanie DNA, čo sa dá popísať aj ako určovanie či rozoberanie, alebo v terminológii úložísk – čítanie. Ide pri tom o určenie toho, aké poradie jednotlivé nukleotidy, teda písmená v samostatnom reťazci DNA, majú. O čítanie DNA a teda zmapovanie konkrétnej genetickej informácie sme sa snažili už od jej objavenia v roku 1953. Po prvýkrát sa nám to podarilo v roku 1977, kedy sme sekvencovaním zmapovali DNA vírusu Phi X 174, ktorý pozostával z 5386 párov písmen.
Väčšie genómy zložitejších foriem života sme zmapovali po prvýkrát v 90. rokoch minulého storočia, pričom ten ľudský s 3,2 miliardami párov, bol zmapovaný v roku 2007 (veľkosť však nie je všetko, niektoré druhy rastlín a rýb majú genóm viac ako stonásobne väčší). V roku 2013 sa dokonca podarilo zmapovať aj genóm vyhynutého rodu človeka neandertálskeho, ktorý sme extrahovali zo 130 000 rokov starej kosti nájdenej v sibírskej jaskyni.
Postupom času bolo vyvinutých mnoho postupov čítania DNA, pričom jeden z prvých (Maxam-Gilbertova metóda) používal rozoberanie chemikáliami. Kým jeden roztok rozštiepil guanín (G) a odhalil jeho pozíciu v DNA špirále, iný zas rozštiepil cytozín (C) a tymín (T), čím odhalil zas ich pozíciu.
Veľký pokrok v rýchlosti sa dosiahol objavením metódy nazývanej ako pyrosekvencovanie, pri ktorom sa reťazec DNA použije ako podklad pre polymerázu, ktorá s ním vytvorí pár. Rozdielom je, že doplnkový pár obsahuje fluorescenčné nukleotidy (alebo laicky povedané písmená iných chemikálií) pričom pri spárovaní vyžiaria špecifický druh svetla. To je možné opticky sledovať a informáciu o písmenách pôvodného reťazca tak čítať.
Ako predošlé riadky napovedajú, pri čítaní DNA rozoberáme, a teda výslednú informáciu čítaním ničíme. To na oko môže vyzerať ako katastrofický problém, avšak takýto fakt je technicky v počítačoch riešený rutinne. Napríklad operačná pamäť v podobe DDR4 stráca svoje hodnoty už za pár milisekúnd, a preto je jej kompletný obsah nutné čítať a znova zapisovať do tranzistorov každých 64 milisekúnd, teda zhruba 15 000-krát za každú sekundu.
Pri používaní počítača tento fakt ale nijako nepociťujeme. V prípade deštruktívneho čítania DNA hrá do karát fakt, že už existujúcu DNA informáciu je možné rýchlo, lacno a energeticky nenáročne mnohonásobne duplikovať, napríklad pomocou Polymerázovej reťazovej reakcie. To znamená, že v rámci úložiska by sme tesne pred deštruktívnym čítaním konkrétnych úsekov mohli dáta opakovane zdvojovať a uchovávať ich.

V roku 2010 bol rekord DNA zápisu len na úrovni 7920 bitov, teda necelý 1 kB
Na zakódovanie binárnej informácie do DNA použili všetky štyri písmená. Nula bola reprezentovaná buď písmenom A alebo C, zatiaľ čo jednotka písmenom G alebo T. To umožnilo informáciu kódovať rôzne a vyhnúť sa kombináciám, ktoré bolo ťažké vytvárať alebo naopak čítať. Napríklad problematický dlhý reťazec vo formáte 0101010101, ktorý pôvodne pozostával z opakovania GCGCGCGCGC, sme tak mohli nahradiť striedavým zápisom GCATGVATGC, čo bolo na výrobu omnoho jednoduchšie a menej chybové. Churchovi a jeho tímu sa podarilo vytvoriť reťazce DNA reprezentujúce Churchovu knihu Regenesis, pojednávajúcu o syntetickej biológii. Kniha bola uložená binárne v jednoduchom HTML formáte, pričom obsahovala 53 426 slov, 11 obrázkov vo formáte JPG a malý javascriptový kód. Spolu šlo o necelý megabajt, presnejšie 658 kB dát. Týchto viac ako päť miliónov bitov biológovia zakódovali do krátkych DNA molekúl a následne úspešne prečítali (z 5,27 milióna bitov bolo zle prečítaných 10, ktoré bolo nutné ručne opraviť).
Aký pokrok sme dosiahli v posledných rokoch a aké vývojové problémy dnes riešime
V posledných piatich rokoch sme sa s vývojom v tomto smere posunuli o značný kus vpred a idea praktického DNA úložiska sa začala čoraz viac zhmotňovať. Vďaka vedeckému výskumu v rámci biológie a iných príbuzných odporov DNA sekvencujeme a syntetizujeme stále rýchlejšie, lacnejšie a v stále väčšom objeme. To sa pre vývoj úložísk nesmierne hodí a mnoho výskumných tímov sa snaží aktuálny pokrok prevziať a doplniť ho o praktické metódy použiteľného dátového zápisu.
Darí sa nám zapisovať čoraz dlhšie reťazce, pričom pomocou rôznych metód dosahujeme na dĺžku 200 až 250 nukleotidov (s veľkou chybovosťou až na 1000 nukleotidov). Takouto dĺžkou obvykle končí len malé množstvo pokusov, avšak výhodou je, že sa nám darí vyrábať ich čoraz viac paralelne, čo mieru neúspechu potláča. Obvyklým postupom je, že začneme strojom dávkovo syntetizovať paralelne veľké množstvo totožných reťazcov, pričom niektoré z nich sa úspešne podarí dokončiť, zatiaľ čo väčšina ostatných v dôsledku chýb zlyhá.
Dá sa to predstaviť ako vytváranie riadkov s veľmi krehkými hrabľami, ktorým sa postupne lámu zuby. Ak začneme robiť riadky v piesku, začíname napríklad s piatimi brázdami a ako kráčame ďalej, zuby hrablí sa lámu a počet riadkov klesá. Avšak v čase keď sa zlomí ten posledný nám zostane jeden dlhý a plynulý riadok. Aj keď je chybovosť v rámci jedného reťazca vysoká, pri spustení mnohých kópií paralelne, sú vždy nejaké perfektné. Trochu to pripomína chybovosť napríklad výrobných procesov polovodičov, pri ktorých je vždy nejaká časť čipov na waferi zlá. Moderné paralelné metódy DNA syntézy umožňujú spúšťať v jednom paralelnom procese až 100 000 rôznych reťazcov, čo nám pomáha dosahovať čoraz lepšie výsledky.
Aj keď sa môže zdať, že dnes bežne dosahované rozmery reťazca DNA sú prikrátke (200 až 250 párov písmen je totiž smiešne číslo v porovnaní s biologickou DNA, ktorá má reťazec dlhý pokojne aj miliardy písmen), v skutočnosti sú pre potreby úložísk dostačujúce. Za rozumné maximum sa dnes považuje 1000 párov, pretože ďalšie predlžovanie rozmeru reťazca už žiadne merateľné výhody neprináša a len spôsobujú problémy ťažšou výrobou a dlhším čítaním nepotrebných dát. Pripomína to problematiku veľkosti sektora HDD/SSD, ktorý takisto používajú logické delenie len na 0,5 alebo 4 kB.
Dôležitým krokom vo vývoji DNA úložísk je takisto riešenie chybovosti zápisu. Prvé pokusy s ukladaním dát riešili chybovosť len veľkou manuálnou fyzickou redundanciou, teda veľkým množstvo rovnakých dát, čo je v reálnom úložisku veľmi nepraktické. V zjednodušenom prirovnaní si to môžeme predstaviť ako uloženie jedného súboru desať či stokrát, pričom následne by sme túto stovku súborov čítali a dúfali, že aspoň jedna kópia je kompletne nepoškodená.
Praktické digitálne úložisko potrebuje chyby zisťovať a riešiť v reálnom čase a na jednom súbore, tak ako to robia tie dnešné typy. Ide o korekciu chýb, vychádzajúcu z kontrolného súčtu, vypočítaného pre každý úsek dát. Tento súčet sa uloží spolu s dátami a môže sa použiť na ich kontrolu a opravu. Zjednodušene si to môžeme ukázať na príklade uloženia štyroch čísiel v podobe 2, 1, 3, 2, ku ktorým uložíme aj ich kontrolný súčet 8. Ak následne dôjde ku chybe pri čítaní a zistia sa dáta 2, 1, CHYBA, 2, úložisko na základe kontrolného súčtu spočíta, že chybové dáta majú hodnotu 3.
Každý typ úložiska vykazuje nejaké percento chýb pri zápise a čítaní, ktoré je treba opravovať. Inak tomu nie je ani pri DNA, pričom v jeho prípade je podiel chýb výrazne naklonený smerom k sekvencovaniu, teda čítaniu, ktoré môže dosiahnuť až na 10 %. Aby logická korekcia podľa kontrolných súčtov mohla dobre fungovať, je potrebné dosiahnuť chybovosť okolo 1 %, s ktorou bežne pracujeme v rámci súčasných magnetických úložísk (ide o surový stav čítania, dáta sa samozrejme v reálnom čase opravujú).
Úspešný pokrok v tomto smere vykonali v roku 2016 výskumníci z laboratória Technicolor Research (Nemecko), ktorí spojili svoje sily s tímom Georgeho Churcha z Harvardovej univerzity a nadviazali na predošlý experiment z roku 2012. Spoločne sa im podarilo po prvýkrát vyvinúť robustnú doprednú metódu korekcie chýb pre DNA zápis, vďaka ktorej úspešne uložili 22 MB dát, teda zhruba 35-krát viac, ako to bolo v predošlom pokuse (číslo vedeckej publikácie doi: 10.1016/j.procs.2016.05.398). Kľúčom bolo ukladanie korekčnej informácie na úplný začiatok každého DNA reťazca, kde je chybovosť najmenšia (náchylnosť na chybu stúpa s jeho dĺžkou). Tým sa dosiahlo minimálnej pravdepodobnosti, že by tieto opravné dáta boli poškodené a mohli sa tak vždy používať na opravu chýb v ďalších častiach reťazca. Chybovosť pri čítaní následne dosiahla úroveň jedného percenta, čiže vďaka účinnej korekcii bolo možné prečítať dáta celkom bez chýb (šlo o krátke video).

Vôbec po prvýkrát došlo ku konštrukcii zariadenia, ktoré rudimentárne pripomínalo úložisko
Zaujímavým prvkom tohto vedeckého projektu je, že vôbec po prvýkrát došlo ku konštrukcii zariadenia, ktoré rudimentárne pripomínalo úložisko, nakoľko obsahovalo všetky potrebné prvky. Pozostávalo zo samostatného syntetizátora DNA, z kontajnera, v ktorom boli jednotlivé reťazce vytvorenej DNA uložené a namapované na jednotku a napokon zo sekvenčného mechanizmu, ktorý DNA reťazce podľa potreby konvertoval späť na digitálne dáta. V predošlých experimentoch sa vykonávalo všetko na rozličných pokročilých biomechanických strojoch, počítačoch a za účasti veľkého množstva ručnej práce.


Kedy DNA úložiská očakávať, ako budú vyzerať a kam sa do dátovej hierarchie zaradia
Úspešné experimenty, ktoré sme v posledných rokoch vykonali, nás napĺňajú optimizmom. Aktuálne riešenia laboratórií Microsoftu a biológov z Washingtonskej univerzity ukazujú, že navrhnuté metódy kódovania, korekcie chýb a spracovávania dát s detekciou konfliktov sú z princípu plne aplikovateľné aj na DNA dátach s objemom niekoľkých TB. V dehydrovaných úložných bunkách (rehydratácia tesne pred čítaním) by pritom mohla byť hustota dát na úrovni jednotiek terabajtov na kubický milimeter, čo je hustota o niekoľko rádov väčšia, aké majú súčasné magnetické úložiská. Budúcnosť sa, pravdaže, veľmi ťažko odhaduje. Obvykle totiž môžeme len primitívne extrapolovať súčasný pokrok a zhodnotiť ho, pričom samozrejme nemôžeme do odhadu zarátať mnoho neznámych faktorov, ktoré skrátka ešte nepoznáme. V konečnom dôsledku tak ide stále len o smelé odhady, ktoré sa nemusia naplniť. Pri pohľade na hmatateľný pokrok, ktorý sme dosiahli v tejto dekáde (1 GB) však veľmi láka povedať, že prvé úspešné experimenty na úrovni zápisu 100 GB (s náhodným čítaním a plnohodnotnou korekciou) sa odohrajú už v nasledujúcich troch rokoch, teda na začiatku tretej dekády. Nebolo by zároveň príliš veľkým prekvapením, ak by sme sa na úroveň niekoľkých terabajtov dokázali posunúť už na jej konci. V prípade, že po roku 2030 začne DNA zápis dosahovať na stovky TB, dostane sa na úroveň iných úložných metód a bude to masívny signál na vyvinutie praktických úložísk tohto typu.
DNA úložiská sa stanú niekedy medzi rokmi 2030 a 2040 prioritným veľkokapacitným dátovým nosičom ľudstva.
Možnostiam škálovania molekulárnych úložísk z hľadiska dátovej hustoty sa totiž tie tranzistorové a magnetické nemôžu nikdy rovnať. Do hry teda vstúpi cena a ak pokles finančných nárokov na syntézu a sekvencovanie DNA bude v nasledujúcej dekáde pokračovať tak ako v tej súčasnej, DNA úložiská sa stanú niekedy medzi rokmi 2030 a 2040 prioritným veľkokapacitným dátovým nosičom ľudstva.


Podobné články

Garmin v tichosti pridal nové funkcie. Aktivujte si ich

Garmin Index Sleep Monitor: Riešenie pre tých, čo nechcú spať s hodinkami (RECENZIA)

Ako fungujú upozornenia na vysoký krvný tlak pomocou Apple Watch

BMW iX3 Neue Klasse: štyri supermozgy, radosť z jazdy a nadčasový dizajn

Apple rozšíril nástroje na ochranu detí a tínedžerov na internete

Chaos v aplikáciách Windows 11: Copilot sa nainštaluje automaticky


